Я провел так много лет, используя и передавая свою любовь к R и используя Python минимально. Недавно прочитав о машинном обучении в Python, я решил заняться забавным небольшим ML-проектом, используя Python от начала до конца.
Ниже следует использование аккуратного набора данных из хранилища машинного обучения UCI . Данные содержат результаты теста по математике 649 учеников в 2 португальских школах. Что хорошо в этом наборе данных, так это то, что в дополнение к оценкам учащихся по 3 математике, им также удалось собрать целый набор демографических переменных (и некоторых поведенческих). Это привело меня к вопросу о том, насколько хорошо вы можете предсказать итоговый результат теста по математике, основываясь только на демографии и поведении. Другими словами, кто, вероятно, преуспеет, и кто, вероятно, танк?
Я должен признать, прежде чем я продолжу, я изначально намеревался провести этот анализ только на Python, но на самом деле я чувствовал себя потерянным на три четверти пути и просто выполнил всю проклятую вещь в R. Как только я закончил анализ в R к своему После этого я вернулся к анализу Python и продолжал, пока не добился своего разумного удовлетворения. По этой причине на каждом этапе анализа я покажу вам код, который я использовал в Python, результаты, а затем то же самое в R. Не рассматривайте это как сравнение возможностей машинного обучения Python против R как такового. Пожалуйста, воспринимайте это как сравнение моего понимания того, как проводить машинное обучение в Python против R!
Без дальнейших церемоний, давайте начнем с некоторых операторов импорта в Python и операторов библиотеки в R:
#Python Code
from pandas import *
from matplotlib import *
import seaborn as sns
sns.set_style("darkgrid")
import matplotlib.pyplot as plt
%matplotlib inline # I did this in ipython notebook, this makes the graphs show up inline in the notebook.
import statsmodels.formula.api as smf
from scipy import stats
from numpy.random import uniform
from numpy import arange
from sklearn.ensemble import RandomForestRegressor
from sklearn.metrics import mean_squared_error
from math import sqrt
mat_perf = read_csv('/home/inkhorn/Student Performance/student-mat.csv', delimiter=';')
Я хотел бы прокомментировать количество операторов импорта, которые я написал в этом скрипте Python. Одиннадцать !! Это даже нормально? Обратите внимание на меньшее количество операторов библиотеки в моем блоке кода R ниже:
#R Code
library(ggplot2)
library(dplyr)
library(ggthemr)
library(caret)
ggthemr('flat') # I love ggthemr!
mat_perf = read.csv('student-mat.csv', sep = ';')
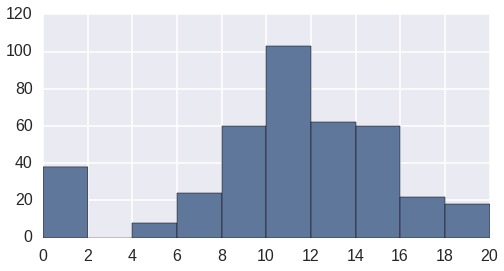
Теперь давайте сделаем краткий график нашей целевой переменной — баллов по итоговому тесту по математике для студентов под названием «G3».
#Python Code
sns.set_palette("deep", desat=.6)
sns.set_context(context='poster', font_scale=1)
sns.set_context(rc={"figure.figsize": (8, 4)})
plt.hist(mat_perf.G3)
plt.xticks(range(0,22,2))
Распределение итоговых тестов по математике («G3»)

Это выглядит довольно приятно для моих глаз. Теперь давайте посмотрим код для того же самого в R (я знаю, визуальная тема отличается. Так что подайте в суд на меня!)
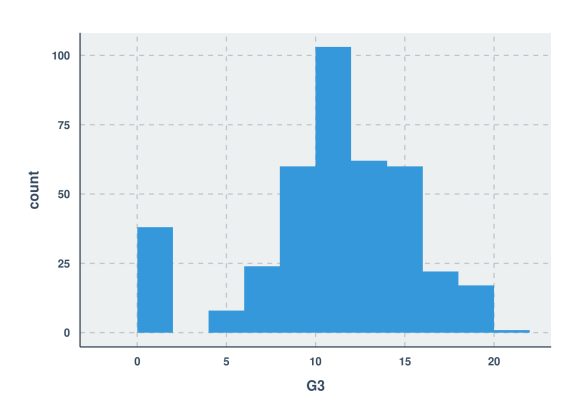
#R Code
ggplot(mat_perf) + geom_histogram(aes(x=G3), binwidth=2)
Вы заметите, что мне не нужно настраивать параметры палитры или размера шрифта для графика R, потому что я использовал очень забавный пакет ggthemr . Вы выбираете нужную визуальную тему, объявляете ее на ранней стадии, и тогда все последующие сюжеты будут иметь одинаковую тему! Однако я скрыл команду, изменяющую высоту и ширину фигуры. Я установил размер рисунка с помощью rmarkdown, иначе я бы просто изменил размер вручную, используя меню экспорта в рамке графика в RStudio. Я думаю, что оба графика выглядят довольно хорошо, хотя я очень неравнодушен к работе с ggthemr!
Однофакторные оценки значения переменных для выбора признаков
Ниже, что я сделал на обоих языках, чтобы циклически перебирал каждую переменную в наборе данных (исключая предыдущие результаты тестов), вставлял имя переменной в словарь / список и получал меру важности того, насколько прогнозирующей является эта переменная, отдельно, итоговой оценки по математике (переменная G3). Конечно, если переменная является качественной, тогда я получаю F балл от ANOVA, и если она количественная, то я получаю балл по регрессии.
В случае Python это достигается в обоих случаях с помощью функции ols из пакета statsmodels scipy. В случае R я добился этого, используя функцию aov для качественной и функцию lm для количественных переменных. Численный результат, как вы увидите из графиков, тот же.
#Python Code
test_stats = {'variable': [], 'test_type' : [], 'test_value' : []}
for col in mat_perf.columns[:-3]:
test_stats['variable'].append(col)
if mat_perf[col].dtype == 'O':
# Do ANOVA
aov = smf.ols(formula='G3 ~ C(' + col + ')', data=mat_perf, missing='drop').fit()
test_stats['test_type'].append('F Test')
test_stats['test_value'].append(round(aov.fvalue,2))
else:
# Do correlation
print col + '\n'
model = smf.ols(formula='G3 ~ ' + col, data=mat_perf, missing='drop').fit()
value = round(model.tvalues[1],2)
test_stats['test_type'].append('t Test')
test_stats['test_value'].append(value)
test_stats = DataFrame(test_stats)
test_stats.sort(columns='test_value', ascending=False, inplace=True)
#R Code
test.stats = list(test.type = c(), test.value = c(), variable = c())
for (i in 1:30) {
test.stats$variable[i] = names(mat_perf)[i]
if (is.factor(mat_perf[,i])) {
anova = summary(aov(G3 ~ mat_perf[,i], data=mat_perf))
test.stats$test.type[i] = "F test"
test.stats$test.value[i] = unlist(anova)[7]
}
else {
reg = summary(lm(G3 ~ mat_perf[,i], data=mat_perf))
test.stats$test.type[i] = "t test"
test.stats$test.value[i] = reg$coefficients[2,3]
}
}
test.stats.df = arrange(data.frame(test.stats), desc(test.value))
test.stats.df$variable = reorder(test.stats.df$variable, -test.stats.df$test.value)
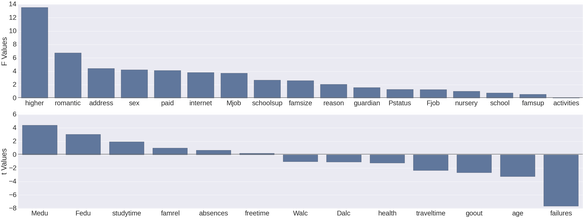
А теперь для графиков. Опять же, вы увидите немного больше кода для графа Python против графа R. Возможно, кто-то сможет показать мне код, который не содержит столько строк, или, может быть, именно так обстоят дела с графикой в Python. Не стесняйтесь обучать меня ?
#Python Code
f, (ax1, ax2) = plt.subplots(2,1, figsize=(48,18), sharex=False)
sns.set_context(context='poster', font_scale=1)
sns.barplot(x='variable', y='test_value', data=test_stats.query("test_type == 'F Test'"), hline=.1, ax=ax1, x_order=[x for x in test_stats.query("test_type == 'F Test'")['variable']])
ax1.set_ylabel('F Values')
ax1.set_xlabel('')
sns.barplot(x='variable', y='test_value', data=test_stats.query("test_type == 't Test'"), hline=.1, ax=ax2, x_order=[x for x in test_stats.query("test_type == 't Test'")['variable']])
ax2.set_ylabel('t Values')
ax2.set_xlabel('')
sns.despine(bottom=True)
plt.tight_layout(h_pad=3)
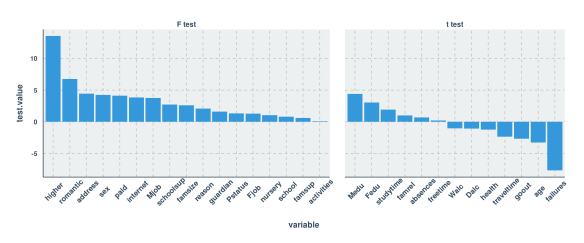
#R Code
ggplot(test.stats.df, aes(x=variable, y=test.value)) +
geom_bar(stat="identity") +
facet_grid(.~test.type , scales="free", space = "free") +
theme(axis.text.x = element_text(angle = 45, vjust=.75, size=11))
Как видите, оценки, которые я генерировал на обоих языках, были, к счастью, одинаковыми. Моей следующей мыслью было использовать только те переменные с тестовым значением (F или t) 3,0 или выше. Ниже вы увидите, что это привело к довольно серьезному снижению прогнозирующей способности по сравнению с либеральным выбором функций.
В действительности, выбор функции, который я использую ниже, вообще не нужен, учитывая размер набора данных в зависимости от количества предикторов и статистический метод, который я использую для прогнозирования оценок (случайный лес). Более того, мой метод выбора функций фактически побудил меня отказаться от определенных переменных, которые позже я нашел важными в моих расширенных моделях! По этой причине было бы неплохо исследовать масштабируемый метод выбора многовариантных объектов (я немного читал о боруте, но скептически отношусь к тому, насколько хорошо он масштабируется), чтобы иметь в своем поясе инструментов. Хватит болтать, и дальше с модельным обучением:
Обучение первой модели случайного леса
#Python code
usevars = [x for x in test_stats.query("test_value >= 3.0 | test_value <= -3.0")['variable']]
mat_perf['randu'] = np.array([uniform(0,1) for x in range(0,mat_perf.shape[0])])
mp_X = mat_perf[usevars]
mp_X_train = mp_X[mat_perf['randu'] <= .67]
mp_X_test = mp_X[mat_perf['randu'] > .67]
mp_Y_train = mat_perf.G3[mat_perf['randu'] <= .67]
mp_Y_test = mat_perf.G3[mat_perf['randu'] > .67]
# for the training set
cat_cols = [x for x in mp_X_train.columns if mp_X_train[x].dtype == "O"]
for col in cat_cols:
new_cols = get_dummies(mp_X_train[col])
new_cols.columns = col + '_' + new_cols.columns
mp_X_train = concat([mp_X_train, new_cols], axis=1)
# for the testing set
cat_cols = [x for x in mp_X_test.columns if mp_X_test[x].dtype == "O"]
for col in cat_cols:
new_cols = get_dummies(mp_X_test[col])
new_cols.columns = col + '_' + new_cols.columns
mp_X_test = concat([mp_X_test, new_cols], axis=1)
mp_X_train.drop(cat_cols, inplace=True, axis=1)
mp_X_test.drop(cat_cols, inplace=True, axis=1)
rf = RandomForestRegressor(bootstrap=True,
criterion='mse', max_depth=2, max_features='auto',
min_density=None, min_samples_leaf=1, min_samples_split=2,
n_estimators=100, n_jobs=1, oob_score=True, random_state=None,
verbose=0)
rf.fit(mp_X_train, mp_Y_train)
После того, как я прошел часть, где я сконструировал наборы для обучения и тестирования (с отфильтрованными «неважными» переменными), я столкнулся с настоящим раздражением. Я узнал, что категориальные переменные должны быть преобразованы в фиктивные переменные перед выполнением моделирования (где каждый уровень категориальной переменной получает свою собственную переменную, содержащую 1 и 0. 1 означает, что уровень присутствовал в этой строке, а 0 означает, что уровень отсутствовал в этом ряду, так называемое «горячее кодирование»). Я полагаю, вы могли бы утверждать, что это требует меньше вычислительных затрат на процедуры моделирования, но когда вы работаете с древовидными ансамблями, я думаю, что это недостаток. Допустим, у вас есть категориальная переменная с 5 функциями, от «а» до «е». Так уж сложилось, что когда вы сравниваете разбивку по той категориальной переменной, где «abc» находится с одной стороны, а «de» — с другой, в зависимой переменной есть очень существенная разница. Как одна горячая кодировка собирается захватить это? И затем, ваш набор данных, который имел определенное количество столбцов, теперь имеет 5 дополнительных столбцов из-за кодировки. «Бла», говорю я!
В любом случае, как вы можете видеть выше, я использовал функцию get_dummies для выполнения горячего кодирования. Кроме того, вы увидите, что я назначил две трети данных для тренировочного набора и одну треть для тестового набора. Теперь давайте посмотрим те же шаги в R:
#R Code
keep.vars = match(filter(test.stats.df, abs(test.value) >= 3)$variable, names(mat_perf))
ctrl = trainControl(method="repeatedcv", number=10, selectionFunction = "oneSE")
mat_perf$randu = runif(395)
test = mat_perf[mat_perf$randu > .67,]
trf = train(mat_perf[mat_perf$randu <= .67,keep.vars], mat_perf$G3[mat_perf$randu <= .67],
method="rf", metric="RMSE", data=mat_perf,
trControl=ctrl, importance=TRUE)
Подожди минуту. Действительно ли я просто обучил модель случайного леса на R, провел перекрестную проверку и подготовил набор данных для тестирования с помощью 5 команд!?!? Это было намного проще, чем делать эти приготовления и не выполнять перекрестную проверку в Python! Я действительно пытался выяснить перекрестную проверку в sklearn, но потом у меня возникли проблемы с доступом к переменным значениям. Мне очень нравится пакет caret ? Далее, давайте посмотрим, как каждая из моделей делала это на своем тестовом наборе:
Тестирование первой модели случайного леса
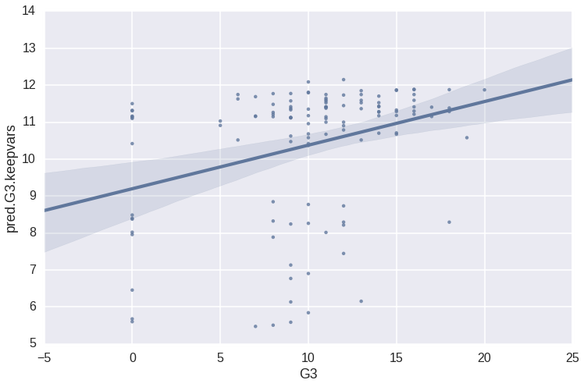
#Python Code
y_pred = rf.predict(mp_X_test)
sns.set_context(context='poster', font_scale=1)
first_test = DataFrame({"pred.G3.keepvars" : y_pred, "G3" : mp_Y_test})
sns.lmplot("G3", "pred.G3.keepvars", first_test, size=7, aspect=1.5)
print 'r squared value of', stats.pearsonr(mp_Y_test, y_pred)[0]**2
print 'RMSE of', sqrt(mean_squared_error(mp_Y_test, y_pred))
R ^ 2 значение 0,104940038879
RMSE 4,66552400292
Здесь, как и во всех случаях при прогнозировании с использованием sklearn, я использую метод прогнозирования для генерации прогнозируемых значений из модели с использованием набора тестирования, а затем строю прогноз («pred.G3.keepvars») против фактических значений (« G3 ″) с помощью функции lmplot. Мне нравится синтаксис, который использует функция lmplot из пакета seaborn, поскольку он прост и знаком мне из мира R (где аргументы состоят из «переменной X, переменной Y, имени набора данных, других эстетических аргументов). Как видно из приведенного выше графика и значения R ^ 2, эта модель отстой. Еще одна вещь, которая мне здесь нравится — это качество графика, который выводит Seaborn. Мило! Это выглядит довольно современно, текст очень читабелен, и ничто не выглядит резким или неровным в сюжете. Хорошо, теперь давайте посмотрим на код и вывод в R, используя те же предикторы.
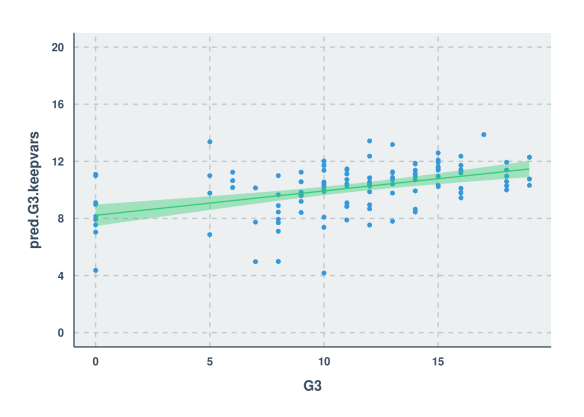
#R Code
test$pred.G3.keepvars = predict(trf, test, "raw")
cor.test(test$G3, test$pred.G3.keepvars)$estimate[[1]]^2
summary(lm(test$G3 ~ test$pred.G3.keepvars))$sigma
ggplot(test, aes(x=G3, y=pred.G3.keepvars)) + geom_point() + stat_smooth(method="lm") + scale_y_continuous(breaks=seq(0,20,4), limits=c(0,20))
R ^ 2 значение 0,198648
RMSE 4,148194
Ну, похоже, эта модель отстой немного меньше, чем модель Python. С точки зрения качества сюжет выглядит очень красиво (спасибо еще раз, ggplot2 и ggthemr!), Хотя по умолчанию параметр альфа не установлен для учета переполнения. Страница документации для ggplot2 предлагает установить альфа = .05, но для этого конкретного набора данных лучше установить его на .5.
Наконец, в этом разделе давайте рассмотрим значения переменных, сгенерированные для каждой модели обучения:
#Python Code
importances = DataFrame({'cols':mp_X_train.columns, 'imps':rf.feature_importances_})
print importances.sort(['imps'], ascending=False)
cols imps
3 failures 0.641898
0 Medu 0.064586
10 sex_F 0.043548
19 Mjob_services 0.038347
11 sex_M 0.036798
16 Mjob_at_home 0.036609
2 age 0.032722
1 Fedu 0.029266
15 internet_yes 0.016545
6 romantic_no 0.013024
7 romantic_yes 0.011134
5 higher_yes 0.010598
14 internet_no 0.007603
4 higher_no 0.007431
12 paid_no 0.002508
20 Mjob_teacher 0.002476
13 paid_yes 0.002006
18 Mjob_other 0.001654
17 Mjob_health 0.000515
8 address_R 0.000403
9 address_U 0.000330
#R Code
varImp(trf)
## rf variable importance
##
## Overall
## failures 100.000
## romantic 49.247
## higher 27.066
## age 17.799
## Medu 14.941
## internet 12.655
## sex 8.012
## Fedu 7.536
## Mjob 5.883
## paid 1.563
## address 0.000
Мое первое наблюдение заключается в том, что мне было намного проще получить значения переменных в R, чем в Python. Далее вы наверняка увидите симптом фиктивного кодирования, который я должен был сделать для категориальных переменных. Это не весело, но мы будем переживать этот пример анализа, верно? Теперь давайте посмотрим, какие переменные оказались на вершине:
Принимая во внимание, что ошибки, уровень образования матери, пол и работа матери заняли верхнюю строчку списка для модели Python, первые 4 отличались от неудач в модели R.
С пониманием того, что метод выбора переменных, который я использовал, был неуместен, давайте перейдем к обучению модели случайного леса с использованием всех предикторов, кроме двух предыдущих тестов. Поскольку я уже прокомментировал свои мысли о различных этапах процесса, я прокомментирую только различия в результатах в оставшихся разделах.
Обучение и тестирование второй модели случайного леса
#Python Code
#aav = almost all variables
mp_X_aav = mat_perf[mat_perf.columns[0:30]]
mp_X_train_aav = mp_X_aav[mat_perf['randu'] <= .67]
mp_X_test_aav = mp_X_aav[mat_perf['randu'] > .67]
# for the training set
cat_cols = [x for x in mp_X_train_aav.columns if mp_X_train_aav[x].dtype == "O"]
for col in cat_cols:
new_cols = get_dummies(mp_X_train_aav[col])
new_cols.columns = col + '_' + new_cols.columns
mp_X_train_aav = concat([mp_X_train_aav, new_cols], axis=1)
# for the testing set
cat_cols = [x for x in mp_X_test_aav.columns if mp_X_test_aav[x].dtype == "O"]
for col in cat_cols:
new_cols = get_dummies(mp_X_test_aav[col])
new_cols.columns = col + '_' + new_cols.columns
mp_X_test_aav = concat([mp_X_test_aav, new_cols], axis=1)
mp_X_train_aav.drop(cat_cols, inplace=True, axis=1)
mp_X_test_aav.drop(cat_cols, inplace=True, axis=1)
rf_aav = RandomForestRegressor(bootstrap=True,
criterion='mse', max_depth=2, max_features='auto',
min_density=None, min_samples_leaf=1, min_samples_split=2,
n_estimators=100, n_jobs=1, oob_score=True, random_state=None,
verbose=0)
rf_aav.fit(mp_X_train_aav, mp_Y_train)
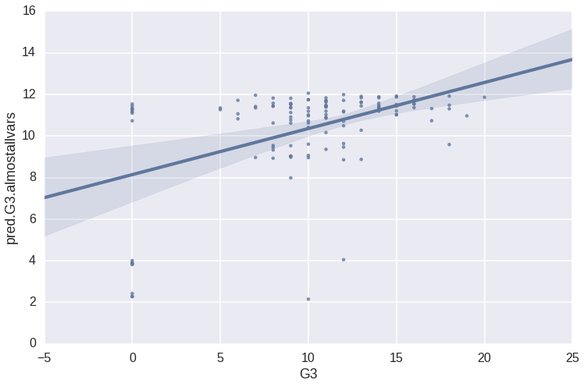
y_pred_aav = rf_aav.predict(mp_X_test_aav)
second_test = DataFrame({"pred.G3.almostallvars" : y_pred_aav, "G3" : mp_Y_test})
sns.lmplot("G3", "pred.G3.almostallvars", second_test, size=7, aspect=1.5)
print 'r squared value of', stats.pearsonr(mp_Y_test, y_pred_aav)[0]**2
print 'RMSE of', sqrt(mean_squared_error(mp_Y_test, y_pred_aav))
R ^ 2 значение 0,226587731888
RMSE 4,3338674965
По сравнению с первой моделью Python R ^ 2 на этой модели более чем вдвое выше (первый R ^ 2 был .10494), а RMSE ниже на 7,1% (первая была 4.6666254). Предсказанный против фактического графика подтверждает, что прогнозы все еще не выглядят фантастическими по сравнению с фактическими данными, что, вероятно, является основной причиной, по которой среднеквадратическое среднеквадратичное отклонение не уменьшилось так сильно. Теперь к коду R используем те же предикторы:
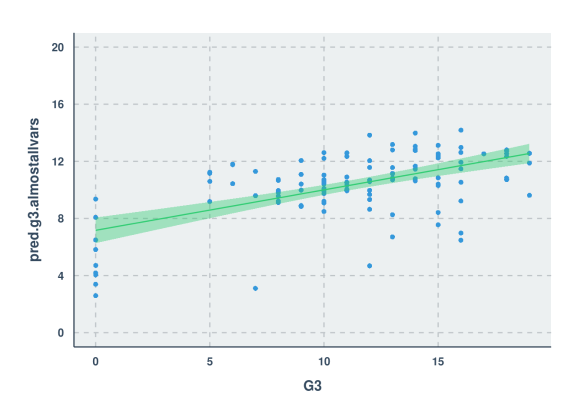
#R code
trf2 = train(mat_perf[mat_perf$randu <= .67,1:30], mat_perf$G3[mat_perf$randu <= .67],
method="rf", metric="RMSE", data=mat_perf,
trControl=ctrl, importance=TRUE)
test$pred.g3.almostallvars = predict(trf2, test, "raw")
cor.test(test$G3, test$pred.g3.almostallvars)$estimate[[1]]^2
summary(lm(test$G3 ~ test$pred.g3.almostallvars))$sigma
ggplot(test, aes(x=G3, y=pred.g3.almostallvars)) + geom_point() +
stat_smooth() + scale_y_continuous(breaks=seq(0,20,4), limits=c(0,20))
R ^ 2 значение 0,3262093
СКО 3,8037318
По сравнению с первой моделью R, R ^ 2 на этой примерно в 1,64 раза выше (первый R ^ 2 был .19865), а RMSE ниже на 8,3% (первая была 4,148194). Хотя эта конкретная модель действительно лучше предсказывает значения в наборе тестов, чем та, которая построена в Python с использованием тех же переменных, я все же не решусь предположить, что этот процесс по своей природе лучше для этого набора данных. Из-за случайности, присущей случайным лесам, один прогон обучения может быть достаточно удачным для получения результатов, подобных описанным выше, тогда как в других случаях результаты могут быть даже несколько хуже, чем то, что мне удалось получить в Python. Я подтвердил это, и на самом деле большинство дополнительных прогонов этой модели в R, по-видимому, приводили к R ^ 2 около 0,20 и RMSE около 4,2.
Опять же, давайте посмотрим на переменные значения, генерируемые для каждой модели обучения:
#Python Code
importances_aav = DataFrame({'cols':mp_X_train_aav.columns, 'imps':rf_aav.feature_importances_})
print importances_aav.sort(['imps'], ascending=False)
cols imps
5 failures 0.629985
12 absences 0.057430
1 Medu 0.037081
41 schoolsup_yes 0.036830
0 age 0.029672
23 Mjob_at_home 0.029642
16 sex_M 0.026949
15 sex_F 0.026052
40 schoolsup_no 0.019097
26 Mjob_services 0.016354
55 romantic_yes 0.014043
51 higher_yes 0.012367
2 Fedu 0.011016
39 guardian_other 0.010715
37 guardian_father 0.006785
8 goout 0.006040
11 health 0.005051
54 romantic_no 0.004113
7 freetime 0.003702
3 traveltime 0.003341
#R Code
varImp(trf2)
## rf variable importance
##
## only 20 most important variables shown (out of 30)
##
## Overall
## absences 100.00
## failures 70.49
## schoolsup 47.01
## romantic 32.20
## Pstatus 27.39
## goout 26.32
## higher 25.76
## reason 24.02
## guardian 22.32
## address 21.88
## Fedu 20.38
## school 20.07
## traveltime 20.02
## studytime 18.73
## health 18.21
## Mjob 17.29
## paid 15.67
## Dalc 14.93
## activities 13.67
## freetime 12.11
Теперь в обоих случаях мы видим, что пропуски и неудачи считаются двумя наиболее важными переменными для прогнозирования итоговой оценки по математике. Это имеет смысл для меня, но, откровенно говоря, немного грустно, что две наиболее важные переменные настолько отрицательны ? Перейдем к третьей модели случайного леса, содержащей все, начиная со второй, с добавлением оценок студентов на втором экзамене по математике. !
Обучение и тестирование третьей модели случайного леса
#Python Code
#allv = all variables (except G1)
allvars = range(0,30)
allvars.append(31)
mp_X_allv = mat_perf[mat_perf.columns[allvars]]
mp_X_train_allv = mp_X_allv[mat_perf['randu'] <= .67]
mp_X_test_allv = mp_X_allv[mat_perf['randu'] > .67]
# for the training set
cat_cols = [x for x in mp_X_train_allv.columns if mp_X_train_allv[x].dtype == "O"]
for col in cat_cols:
new_cols = get_dummies(mp_X_train_allv[col])
new_cols.columns = col + '_' + new_cols.columns
mp_X_train_allv = concat([mp_X_train_allv, new_cols], axis=1)
# for the testing set
cat_cols = [x for x in mp_X_test_allv.columns if mp_X_test_allv[x].dtype == "O"]
for col in cat_cols:
new_cols = get_dummies(mp_X_test_allv[col])
new_cols.columns = col + '_' + new_cols.columns
mp_X_test_allv = concat([mp_X_test_allv, new_cols], axis=1)
mp_X_train_allv.drop(cat_cols, inplace=True, axis=1)
mp_X_test_allv.drop(cat_cols, inplace=True, axis=1)
rf_allv = RandomForestRegressor(bootstrap=True,
criterion='mse', max_depth=2, max_features='auto',
min_density=None, min_samples_leaf=1, min_samples_split=2,
n_estimators=100, n_jobs=1, oob_score=True, random_state=None,
verbose=0)
rf_allv.fit(mp_X_train_allv, mp_Y_train)
y_pred_allv = rf_allv.predict(mp_X_test_allv)
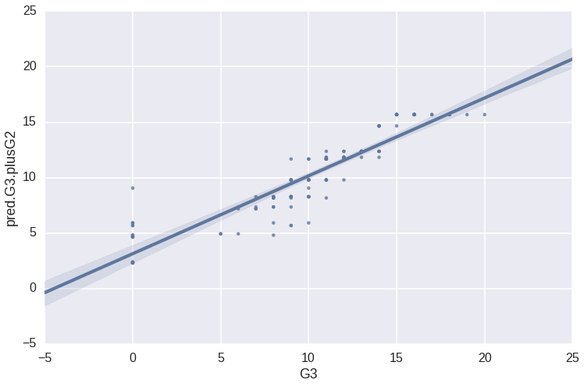
third_test = DataFrame({"pred.G3.plusG2" : y_pred_allv, "G3" : mp_Y_test})
sns.lmplot("G3", "pred.G3.plusG2", third_test, size=7, aspect=1.5)
print 'r squared value of', stats.pearsonr(mp_Y_test, y_pred_allv)[0]**2
print 'RMSE of', sqrt(mean_squared_error(mp_Y_test, y_pred_allv))
R ^ 2 значение 0,836089929903
RMSE 2,11895794845
Очевидно, что мы добавили весьма прогнозируемую информацию здесь, добавив оценки от их второго экзамена по математике (имя переменной было «G2»). Сначала я неохотно добавлял эту переменную, потому что, когда вы прогнозируете тестовые отметки с помощью предыдущих тестовых отметок, тогда это препятствует тому, чтобы модель была полезной намного раньше в том году, когда эти тесты не проводились. Тем не менее, я действительно хотел посмотреть, как будет выглядеть модель, когда я ее все равно включу! Теперь давайте посмотрим, насколько прогнозирующими были эти переменные, когда я поместил их в модель в R:
#R Code
trf3 = train(mat_perf[mat_perf$randu <= .67,c(1:30,32)], mat_perf$G3[mat_perf$randu <= .67],
method="rf", metric="RMSE", data=mat_perf,
trControl=ctrl, importance=TRUE)
test$pred.g3.plusG2 = predict(trf3, test, "raw")
cor.test(test$G3, test$pred.g3.plusG2)$estimate[[1]]^2
summary(lm(test$G3 ~ test$pred.g3.plusG2))$sigma
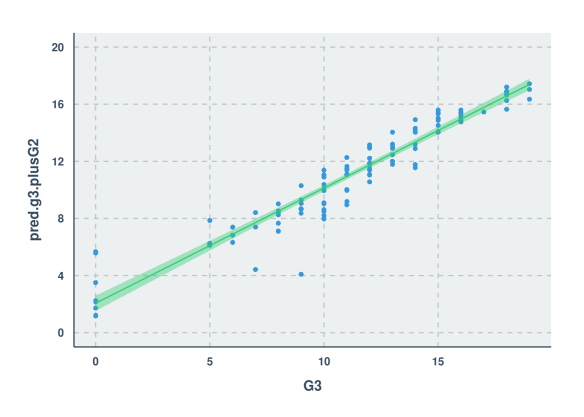
ggplot(test, aes(x=G3, y=pred.g3.plusG2)) + geom_point() +
stat_smooth(method="lm") + scale_y_continuous(breaks=seq(0,20,4), limits=c(0,20))
R ^ 2 значение 0,9170506
RMSE 1,3336087
Что ж, похоже, что снова у нас есть случай, когда модель R оказалась лучше, чем модель Python. Я нахожу примечательным, что когда вы смотрите на график рассеяния для модели Python, вы можете видеть, как выглядят шаги в точках, когда вы сканируете свои глаза от нижней левой части линии тренда до верхней правой части. Похоже, что модель случайного леса в R выиграла от процесса настройки, и в результате распределение остатков более гомоскедастично и, очевидно, ближе к линии регрессии, чем модель Python. Мне все еще интересно, насколько похожими были бы эти результаты, если бы я выполнил анализ Python, настроившись при перекрестной проверке, как в R!
В последний раз давайте рассмотрим значения переменных, сгенерированные для каждой модели обучения:
#Python Code
importances_allv = DataFrame({'cols':mp_X_train_allv.columns, 'imps':rf_allv.feature_importances_})
print importances_allv.sort(['imps'], ascending=False)
cols imps
13 G2 0.924166
12 absences 0.075834
14 school_GP 0.000000
25 Mjob_health 0.000000
24 Mjob_at_home 0.000000
23 Pstatus_T 0.000000
22 Pstatus_A 0.000000
21 famsize_LE3 0.000000
20 famsize_GT3 0.000000
19 address_U 0.000000
18 address_R 0.000000
17 sex_M 0.000000
16 sex_F 0.000000
15 school_MS 0.000000
56 romantic_yes 0.000000
27 Mjob_services 0.000000
11 health 0.000000
10 Walc 0.000000
9 Dalc 0.000000
8 goout 0.000000
#R Code
varImp(trf3)
## rf variable importance
##
## only 20 most important variables shown (out of 31)
##
## Overall
## G2 100.000
## absences 33.092
## failures 9.702
## age 8.467
## paid 7.591
## schoolsup 7.385
## Pstatus 6.604
## studytime 5.963
## famrel 5.719
## reason 5.630
## guardian 5.278
## Mjob 5.163
## school 4.905
## activities 4.532
## romantic 4.336
## famsup 4.335
## traveltime 4.173
## Medu 3.540
## Walc 3.278
## higher 3.246
Теперь это ОЧЕНЬ говорит, и дает мне представление о том, почему график рассеяния из модели Python имел такое качество лестницы. Модель R учитывает больше переменных, чем модель Python. Очевидно, что G2 выигрывает в обеих моделях, но я полагаю, что он настолько омрачил все остальное в модели Python, что по какой-то причине он просто не нашел применения для какой-либо другой переменной, кроме отсутствий.
Вывод
Это было весело! Для всей работы, которую я делал в Python, я использовал IPython Notebook. Будучи заядлым пользователем RStudio, я не привык к интерактивному кодированию на основе веб-браузера, как, например, в IPython Notebook. Я обнаружил, что мне это нравится, и нашел, что это полезно для размещения информации, которую я использовал для написания этого поста в блоге (по той же причине я также изложил R-часть этого анализа в RMarkdown). Что мне не понравилось в IPython Notebook, так это то, что когда вы закрываете его / закрываете / затем снова его инициализируете, все объекты, которые формируют ваши данные и анализ, исчезают, и все, что у вас осталось, — это результаты. Затем вы должны перезапустить весь ваш код, чтобы ваши объекты снова находились в памяти. Было бы неплохо иметь какую-то удобную функцию для сохранения всего на диск, чтобы вы могли перезагрузить его позже.
Я часто спотыкался, пытаясь выяснить, какие пакеты Python использовать для каждой конкретной цели, и я был легко разочарован. Я должен был постоянно напоминать себе, что это кривая обучения в той же степени, как и для меня, пока я учил R. Это разочарование не должно быть сдерживающим фактором для того, чтобы поднять его и научиться делать машинное обучение в Python. Другая часть моего разочарования была в том, что я не мог получить переменные значения из моих моделей Random Forest в Python, когда я строил их с использованием перекрестной проверки и поиска по сетке. Если у вас есть ссылка, чтобы поделиться со мной, которая показывает пример этого, я был бы рад прочитать ее.
Мне понравился seaborn, и я думаю, что если я потрачу на него больше времени, то, возможно, он послужит достойной альтернативой графике в ggplot2. При этом я потратил так много времени на использование ggplot2, что иногда мне интересно, есть ли что-нибудь, что может сравниться с его гибкостью и элегантностью!
Проблема, которую я упомянул выше с категориальными переменными, раздражает, и это действительно заставляет меня задаться вопросом, будет ли использование модели R на основе дерева по своей сути лучше, потому что она автоматически обрабатывает категориальные переменные по сравнению с Python, где вам нужно горячо кодировать эти переменные.
В общем, я надеюсь, что это было так же полезно и полезно для вас, как и для меня. Важно выходить за пределы своей зоны комфорта время от времени ?