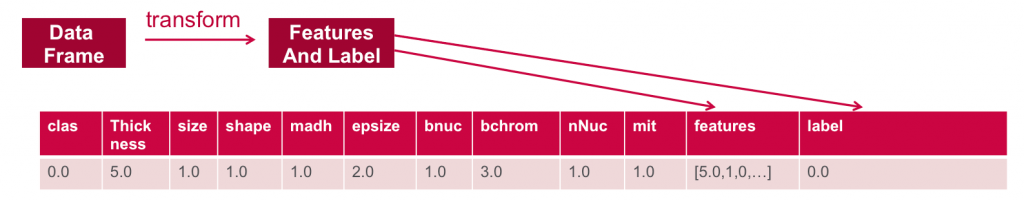
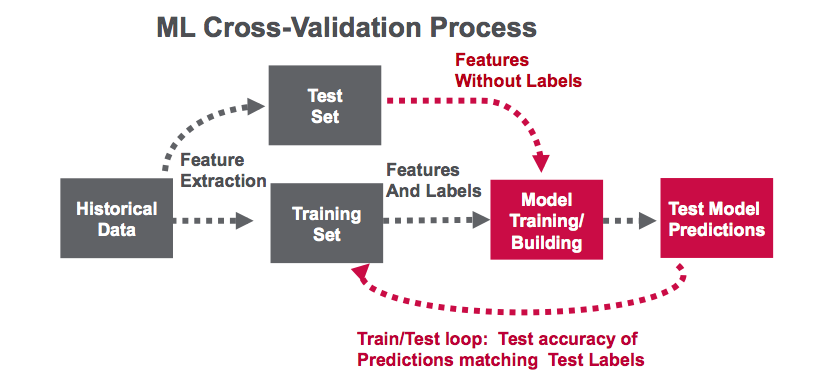
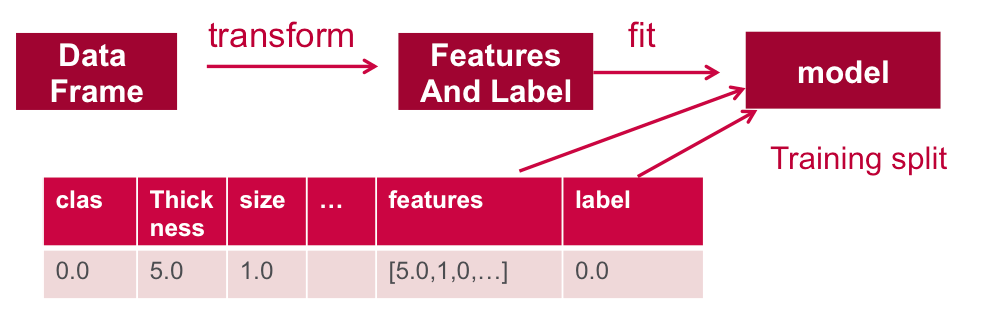
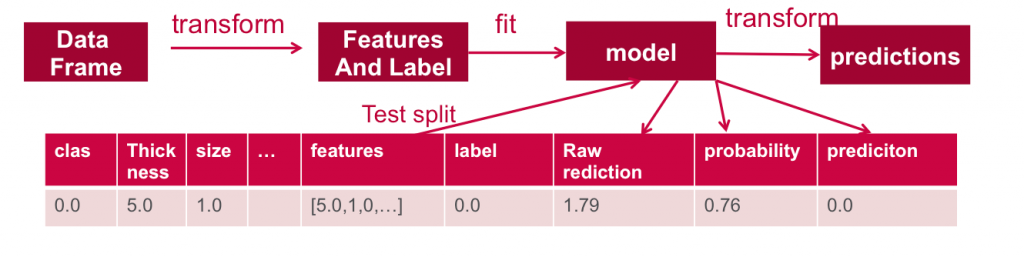
В этом посте я помогу вам начать использовать логистическую регрессию Apache Spark spark.ml для прогнозирования злокачественных новообразований.
Цель библиотеки Spark spark.ml — предоставить набор API поверх DataFrames, которые помогают пользователям создавать и настраивать рабочие процессы или конвейеры машинного обучения. Использование spark.ml с DataFrames повышает производительность за счет интеллектуальной оптимизации.
классификация
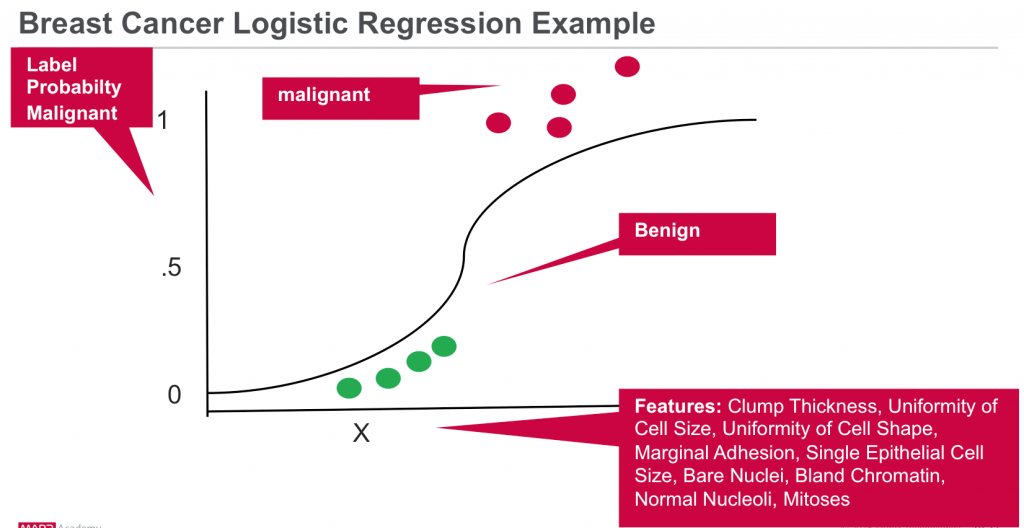
Классификация — это семейство контролируемых алгоритмов машинного обучения, которые определяют, к какой категории относится элемент (например, является ли наблюдение злокачественной ткани злокачественным или нет), на основе помеченных примеров известных предметов (например, наблюдения, известные как злокачественные или нет). ). Классификация берет набор данных с известными метками и предопределенными функциями и изучает, как маркировать новые записи на основе этой информации. Особенности — это «если вопросы», которые вы задаете. Этикетка является ответом на эти вопросы. В приведенном ниже примере, если он ходит, плавает и крякает, как утка, тогда на этикетке будет написано «утка».
Давайте рассмотрим пример наблюдения ткани рака:
- Что мы пытаемся предсказать?
- Является ли наблюдение за образцом злокачественным или нет.
- Это Метка: злокачественная или нет.
- Какие «если вопросы» или свойства, которые вы можете использовать для прогнозирования?
- Характеристики образца ткани: толщина скопления, однородность размера клеток, однородность формы клеток, предельная адгезия, размер единичных эпителиальных клеток, голые ядра, мягкий хроматин, нормальные ядра, митозы.
- Это особенности. Чтобы построить модель классификатора, вы извлекаете интересующие элементы, которые наиболее способствуют классификации.
Логистическая регрессия
Логистическая регрессия является популярным методом прогнозирования двоичного ответа. Это особый случай обобщенных линейных моделей, который предсказывает вероятность результата. Логистическая регрессия измеряет отношения между Y «Метка» и X «Особенности» путем оценки вероятностей с использованием логистической функции. Модель прогнозирует вероятность, которая используется для прогнозирования класса метки.
Проанализируйте наблюдения рака с помощью сценария машинного обучения
Наши данные взяты из Висконсинского диагностического набора данных по раку молочной железы (WDBC), который классифицирует случаи опухоли молочной железы как доброкачественные или злокачественные на основе 9 признаков для прогнозирования диагноза. Для каждого наблюдения рака у нас есть следующая информация:
|
01
02
03
04
05
06
07
08
09
10
11
|
1. Sample code number: id number 2. Clump Thickness: 1 - 103. Uniformity of Cell Size: 1 - 104. Uniformity of Cell Shape: 1 - 105. Marginal Adhesion: 1 - 106. Single Epithelial Cell Size: 1 - 107. Bare Nuclei: 1 - 108. Bland Chromatin: 1 - 109. Normal Nucleoli: 1 - 1010. Mitoses: 1 - 1011. Class: (2 for benign, 4 for malignant) |
CSV-файл «Наблюдение за раком» имеет следующий формат:
|
1
2
3
|
1000025,5,1,1,1,2,1,3,1,1,21002945,5,4,4,5,7,10,3,2,1,21015425,3,1,1,1,2,2,3,1,1,2 |
В этом сценарии мы создадим модель логистической регрессии, чтобы предсказать метку / классификацию злокачественных или не основанную на следующих особенностях:
- Метка → злокачественная или доброкачественная (1 или 0)
- Особенности → {Толщина комков, однородность размера клеток, однородность формы клеток, предельная адгезия, размер единичных эпителиальных клеток, оголенные ядра, мягкий хроматин, нормальные ядра, митозы}
Spark ML предоставляет унифицированный набор высокоуровневых API, созданных поверх DataFrames. Основные понятия в Spark ML:
- DataFrame: ML API использует DataFrames из Spark SQL в качестве набора данных ML.
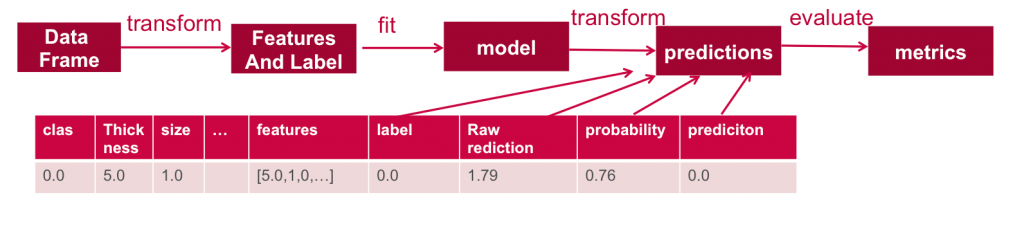
- Transformer: Transformer — это алгоритм, который преобразует один DataFrame в другой DataFrame. Например, превращение DataFrame с функциями в DataFrame с предсказаниями.
- Оценщик: Оценщик — это алгоритм, который может быть помещен в DataFrame для создания Transformer. Например, обучение / настройка на DataFrame и создание модели.
- Конвейер: Конвейер объединяет несколько Трансформаторов и Оценщиков, чтобы определить рабочий процесс ML.
- ParamMaps: параметры для выбора, иногда называемые «сеткой параметров» для поиска.
- Оценщик: Метрика, позволяющая измерить, насколько хорошо подобранная Модель справляется с данными, проведенными в течение длительного времени.
- CrossValidator: Определяет лучший ParamMap и переоснащает Оценщик, используя лучший ParamMap и весь набор данных.
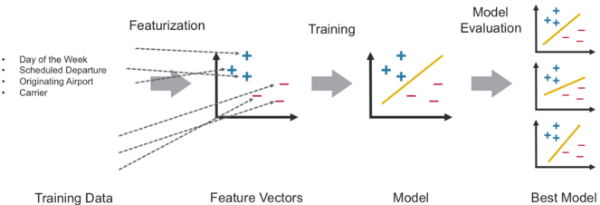
В этом примере будет использоваться рабочий процесс Spark ML, показанный ниже: 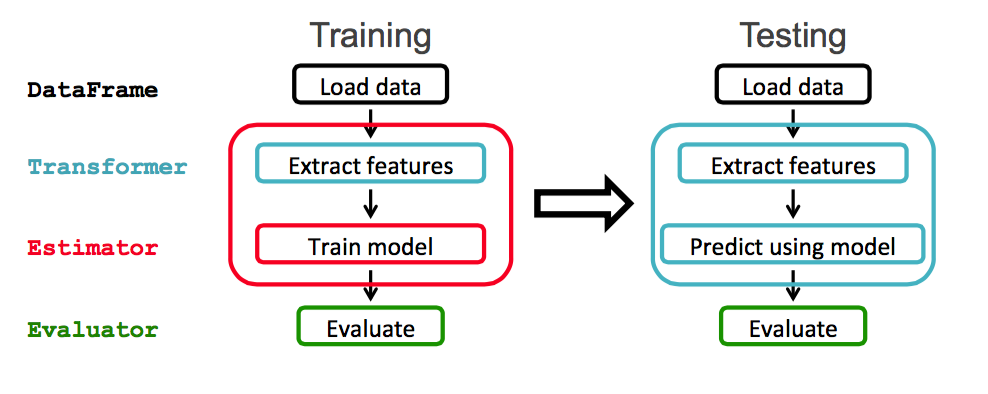
Программного обеспечения
Этот учебник будет работать на Spark 1.6.1
- Вы можете скачать код и данные для запуска этих примеров здесь: https://github.com/caroljmcdonald/spark-ml-lr-cancer
- Примеры в этом посте можно запустить в оболочке Spark после запуска с помощью команды spark-shell.
- Вы также можете запустить код как отдельное приложение, как описано в руководстве по началу работы с Spark в MapR Sandbox .
Войдите в MapR Sandbox, как описано в разделе Начало работы с Spark в MapR Sandbox , используя идентификатор пользователя user01, пароль mapr. Скопируйте файл примера данных в домашнюю директорию песочницы / user / user01 с помощью scp. (Обратите внимание, что вам может потребоваться обновить версию Spark на вашей песочнице) Запустите оболочку Spark с помощью:
|
1
|
$spark-shell --master local[1] |
Загрузка и анализ данных из файла CSV
Сначала мы импортируем пакеты машинного обучения.
(В полях кода комментарии отображаются зеленым, а вывод — синим)
|
01
02
03
04
05
06
07
08
09
10
11
12
13
14
15
|
import org.apache.spark._import org.apache.spark.rdd.RDDimport org.apache.spark.sql.SQLContextimport org.apache.spark.ml.feature.StringIndexerimport org.apache.spark.ml.feature.VectorAssemblerimport org.apache.spark.ml.classification.BinaryLogisticRegressionSummaryimport org.apache.spark.ml.evaluation.BinaryClassificationEvaluatorimport org.apache.spark.ml.classification.LogisticRegressionimport org.apache.spark.ml.feature.StringIndexerimport org.apache.spark.ml.feature.VectorAssemblerimport sqlContext.implicits._import sqlContext._import org.apache.spark.sql.functions._import org.apache.spark.mllib.linalg.DenseVectorimport org.apache.spark.mllib.evaluation.BinaryClassificationMetrics |
Мы используем класс case Scala для определения схемы, соответствующей строке в файле данных csv.
|
1
2
|
// define the Cancer Observation Schemacase class Obs(clas: Double, thickness: Double, size: Double, shape: Double, madh: Double, epsize: Double, bnuc: Double, bchrom: Double, nNuc: Double, mit: Double) |
Приведенные ниже функции анализируют строку из файла данных в классе наблюдения рака.
|
01
02
03
04
05
06
07
08
09
10
|
// function to create a Obs class from an Array of Double.Class Malignant 4 is changed to 1def parseObs(line: Array[Double]): Obs = { Obs( if (line(9) == 4.0) 1 else 0, line(0), line(1), line(2), line(3), line(4), line(5), line(6), line(7), line(8) )}// function to transform an RDD of Strings into an RDD of Double, filter lines with ?, remove first columndef parseRDD(rdd: RDD[String]): RDD[Array[Double]] = { rdd.map(_.split(",")).filter(_(6) != "?").map(_.drop(1)).map(_.map(_.toDouble))} |
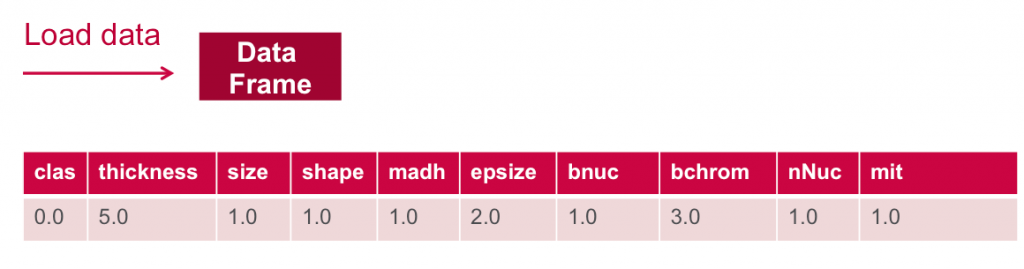
Ниже мы загружаем данные из файла csv в RDD из строк. Затем мы используем преобразование карты для rdd, которое будет применять функцию ParseRDD для преобразования каждого элемента String в RDD в массив Double. Затем мы используем другое преобразование карты, которое будет применять функцию ParseObs для преобразования каждого массива Double в RDD в массив Array of Cancer Observation. Метод toDF () преобразует СДР массива [[Наблюдение за раком]] в Dataframe со схемой класса Наблюдение за раком.
|
1
2
3
4
5
|
// load the data into a DataFrameval rdd = sc.textFile("data/breast_cancer_wisconsin_data.txt")val obsRDD = parseRDD(rdd).map(parseObs)val obsDF = obsRDD.toDF().cache()obsDF.registerTempTable("obs") |
DataFrame printSchema () Печатает схему на консоли в древовидном формате
|
01
02
03
04
05
06
07
08
09
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
|
// Return the schema of this DataFrameobsDF.printSchemaroot |-- clas: double (nullable = false) |-- thickness: double (nullable = false) |-- size: double (nullable = false) |-- shape: double (nullable = false) |-- madh: double (nullable = false) |-- epsize: double (nullable = false) |-- bnuc: double (nullable = false) |-- bchrom: double (nullable = false) |-- nNuc: double (nullable = false) |-- mit: double (nullable = false)// Display the top 20 rows of DataFrame obsDF.show+----+---------+----+-----+----+------+----+------+----+---+|clas|thickness|size|shape|madh|epsize|bnuc|bchrom|nNuc|mit|+----+---------+----+-----+----+------+----+------+----+---+| 0.0| 5.0| 1.0| 1.0| 1.0| 2.0| 1.0| 3.0| 1.0|1.0|| 0.0| 5.0| 4.0| 4.0| 5.0| 7.0|10.0| 3.0| 2.0|1.0|| 0.0| 3.0| 1.0| 1.0| 1.0| 2.0| 2.0| 3.0| 1.0|1.0|...+----+---------+----+-----+----+------+----+------+----+---+only showing top 20 rows |
После создания экземпляра DataFrame вы можете запросить его с помощью SQL-запросов. Вот несколько примеров запросов с использованием Scala DataFrame API:
описать вычисляет статистику для столбца толщины, в том числе count, mean, stddev, min и max
|
01
02
03
04
05
06
07
08
09
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
|
// computes statistics for thickness obsDF.describe("thickness").show+-------+------------------+|summary| thickness|+-------+------------------+| count| 683|| mean| 4.44216691068814|| stddev|2.8207613188371288|| min| 1.0|| max| 10.0|+-------+------------------+ // compute the avg thickness, size, shape grouped by clas (malignant or not) sqlContext.sql("SELECT clas, avg(thickness) as avgthickness, avg(size) as avgsize, avg(shape) as avgshape FROM obs GROUP BY clas ").show+----+-----------------+------------------+------------------+|clas| avgthickness| avgsize| avgshape|+----+-----------------+------------------+------------------+| 1.0|7.188284518828452| 6.577405857740586| 6.560669456066946|| 0.0|2.963963963963964|1.3063063063063063|1.4144144144144144|+----+-----------------+------------------+------------------+ // compute avg thickness grouped by clas (malignant or not) obsDF.groupBy("clas").avg("thickness").show+----+-----------------+|clas| avg(thickness)|+----+-----------------+| 1.0|7.188284518828452|| 0.0|2.963963963963964|+----+-----------------+ |
Особенности извлечения
Чтобы построить модель классификатора, вы сначала извлекаете функции, которые наиболее способствуют классификации. В наборе данных о раке данные помечены двумя классами — 1 (злокачественный) и 0 (не злокачественный).
Функции для каждого элемента состоят из полей, показанных ниже:
- Метка → злокачественная: 0 или 1
- Особенности → {«толщина», «размер», «форма», «madh», «epsize», «bnuc», «bchrom», «nNuc», «mit»}
Определить массив объектов
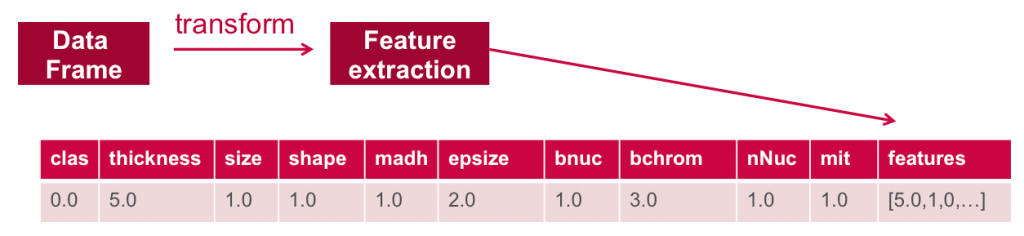
Для того чтобы функции использовались алгоритмом машинного обучения, эти функции преобразуются и помещаются в векторы признаков, которые представляют собой векторы чисел, представляющие значение для каждого объекта.
Ниже VectorAssembler используется для преобразования и возврата нового DataFrame со всеми столбцами объектов в векторном столбце.
|
01
02
03
04
05
06
07
08
09
10
11
12
13
14
15
16
|
//define the feature columns to put in the feature vectorval featureCols = Array("thickness", "size", "shape", "madh", "epsize", "bnuc", "bchrom", "nNuc", "mit")//set the input and output column namesval assembler = new VectorAssembler().setInputCols(featureCols).setOutputCol("features")//return a dataframe with all of the feature columns in a vector columnval df2 = assembler.transform(obsDF)// the transform method produced a new column: features.df2.show+----+---------+----+-----+----+------+----+------+----+---+--------------------+|clas|thickness|size|shape|madh|epsize|bnuc|bchrom|nNuc|mit| features|+----+---------+----+-----+----+------+----+------+----+---+--------------------+| 0.0| 5.0| 1.0| 1.0| 1.0| 2.0| 1.0| 3.0| 1.0|1.0|[5.0,1.0,1.0,1.0,...|| 0.0| 5.0| 4.0| 4.0| 5.0| 7.0|10.0| 3.0| 2.0|1.0|[5.0,4.0,4.0,5.0,...|| 1.0| 8.0|10.0| 10.0| 8.0| 7.0|10.0| 9.0| 7.0|1.0|[8.0,10.0,10.0,8....| |
Затем мы используем StringIndexer для возврата Dataframe со столбцом clas (злокачественный или нет), добавленным в качестве метки.
|
01
02
03
04
05
06
07
08
09
10
11
12
|
// Create a label column with the StringIndexer val labelIndexer = new StringIndexer().setInputCol("clas").setOutputCol("label")val df3 = labelIndexer.fit(df2).transform(df2)// the transform method produced a new column: label.df3.show+----+---------+----+-----+----+------+----+------+----+---+--------------------+-----+|clas|thickness|size|shape|madh|epsize|bnuc|bchrom|nNuc|mit| features|label|+----+---------+----+-----+----+------+----+------+----+---+--------------------+-----+| 0.0| 5.0| 1.0| 1.0| 1.0| 2.0| 1.0| 3.0| 1.0|1.0|[5.0,1.0,1.0,1.0,...| 0.0|| 0.0| 5.0| 4.0| 4.0| 5.0| 7.0|10.0| 3.0| 2.0|1.0|[5.0,4.0,4.0,5.0,...| 0.0|| 0.0| 3.0| 1.0| 1.0| 1.0| 2.0| 2.0| 3.0| 1.0|1.0|[3.0,1.0,1.0,1.0,...| 0.0| |
Ниже данных. Он разделен на набор обучающих данных и набор тестовых данных. 70% данных используются для обучения модели, а 30% будут использоваться для тестирования.
|
1
2
3
|
// split the dataframe into training and test dataval splitSeed = 5043val Array(trainingData, testData) = df3.randomSplit(Array(0.7, 0.3), splitSeed) |
Тренировать модель
Далее мы обучаем модель логистической регрессии с упругой регуляризацией сети
Модель обучается путем установления связей между входными функциями и помеченными выходными данными, связанными с этими функциями.
|
01
02
03
04
05
06
07
08
09
10
|
// create the classifier, set parameters for trainingval lr = new LogisticRegression().setMaxIter(10).setRegParam(0.3).setElasticNetParam(0.8)// use logistic regression to train (fit) the model with the training dataval model = lr.fit(trainingData) // Print the coefficients and intercept for logistic regressionprintln(s"Coefficients: ${model.coefficients} Intercept: ${model.intercept}")Coefficients: (9,[1,2,5,6],[0.06503554553146387,0.07181362361391264,0.07583963853124673,0.0012675057388232965]) Intercept: -1.39319142312609 |
Проверьте модель
Далее мы используем тестовые данные, чтобы получить прогнозы.
|
01
02
03
04
05
06
07
08
09
10
11
12
13
14
|
// run the model on test features to get predictionsval predictions = model.transform(testData) //As you can see, the previous model transform produced a new columns: rawPrediction, probablity and prediction.predictions.show+----+---------+----+-----+----+------+----+------+----+---+--------------------+-----+--------------------+--------------------+----------+|clas|thickness|size|shape|madh|epsize|bnuc|bchrom|nNuc|mit| features|label| rawPrediction| probability|prediction|+----+---------+----+-----+----+------+----+------+----+---+--------------------+-----+--------------------+--------------------+----------+| 0.0| 1.0| 1.0| 1.0| 1.0| 1.0| 1.0| 1.0| 3.0|1.0|[1.0,1.0,1.0,1.0,...| 0.0|[1.17923510971064...|[0.76481024658406...| 0.0|| 0.0| 1.0| 1.0| 1.0| 1.0| 1.0| 1.0| 3.0| 1.0|1.0|[1.0,1.0,1.0,1.0,...| 0.0|[1.17670009823299...|[0.76435395397908...| 0.0|| 0.0| 1.0| 1.0| 1.0| 1.0| 1.0| 1.0| 3.0| 1.0|1.0|[1.0,1.0,1.0,1.0,...| 0.0|[1.17670009823299...|[0.76435395397908...| 0.0|| 0.0| 1.0| 1.0| 1.0| 1.0| 2.0| 1.0| 1.0| 1.0|1.0|[1.0,1.0,1.0,1.0,...| 0.0|[1.17923510971064...|[0.76481024658406...| 0.0|| 0.0| 1.0| 1.0| 1.0| 1.0| 2.0| 1.0| 2.0| 1.0|1.0|[1.0,1.0,1.0,1.0,...| 0.0|[1.17796760397182...|[0.76458217679258...| 0.0|+----+---------+----+-----+----+------+----+------+----+---+--------------------+-----+--------------------+--------------------+----------+ |
Ниже мы оцениваем прогнозы и используем BinaryClassificationEvaluator, который возвращает метрику точности, сравнивая столбец метки теста со столбцом прогноза теста. В этом случае оценка возвращает точность 99%.
|
1
2
3
4
5
6
|
//A common metric used for logistic regression is area under the ROC curve (AUC). We can use the BinaryClasssificationEvaluator to obtain the AUC // create an Evaluator for binary classification, which expects two input columns: rawPrediction and label.val evaluator = new BinaryClassificationEvaluator().setLabelCol("label").setRawPredictionCol("rawPrediction").setMetricName("areaUnderROC")// Evaluates predictions and returns a scalar metric areaUnderROC(larger is better). val accuracy = evaluator.evaluate(predictions) accuracy: Double = 0.9926910299003322 |
Ниже мы рассчитаем еще несколько метрик. Количество ложных и истинных положительных и отрицательных предсказаний также полезно:
- Истинные положительные результаты показывают, как часто модель правильно предсказывала, что опухоль была злокачественной
- Ложные срабатывания показывают, как часто модель предсказывала, что опухоль была злокачественной, когда она была доброкачественной
- Истинные негативы показывают, как модель правильно предсказала, что опухоль была доброкачественной
- Ложные отрицания указывают на то, как часто модель предсказывала, что опухоль была доброкачественной, хотя на самом деле она была злокачественной
|
01
02
03
04
05
06
07
08
09
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
|
// Calculate Metricsval lp = predictions.select( "label", "prediction")val counttotal = predictions.count()val correct = lp.filter($"label" === $"prediction").count()val wrong = lp.filter(not($"label" === $"prediction")).count()val truep = lp.filter($"prediction" === 0.0).filter($"label" === $"prediction").count()val falseN = lp.filter($"prediction" === 0.0).filter(not($"label" === $"prediction")).count()val falseP = lp.filter($"prediction" === 1.0).filter(not($"label" === $"prediction")).count()val ratioWrong=wrong.toDouble/counttotal.toDoubleval ratioCorrect=correct.toDouble/counttotal.toDoublecounttotal: Long = 199correct: Long = 168wrong: Long = 31truep: Long = 128falseN: Long = 30falseP: Long = 1ratioWrong: Double = 0.15577889447236182ratioCorrect: Double = 0.8442211055276382// use MLlib to evaluate, convert DF to RDDval predictionAndLabels =predictions.select("rawPrediction", "label").rdd.map(x => (x(0).asInstanceOf[DenseVector](1), x(1).asInstanceOf[Double]))val metrics = new BinaryClassificationMetrics(predictionAndLabels) println("area under the precision-recall curve: " + metrics.areaUnderPR)println("area under the receiver operating characteristic (ROC) curve : " + metrics.areaUnderROC)// A Precision-Recall curve plots (precision, recall) points for different threshold values, while a receiver operating characteristic, or ROC, curve plots (recall, false positive rate) points. The closer the area Under ROC is to 1, the better the model is making predictions.area under the precision-recall curve: 0.9828385182615946area under the receiver operating characteristic (ROC) curve : 0.9926910299003322 |
Хотите узнать больше?
- Бесплатное обучение Apache по требованию
- MapR и Spark
- Обзор Apache Spark ML
- Apache Spark ML Логистическая регрессия
- Искусственный интеллект читает маммограммы с точностью 99%
В этом посте мы показали, как начать использовать машинное обучение логистической регрессии Apache Spark для классификации. Если у вас есть какие-либо дополнительные вопросы по этому учебнику, пожалуйста, задавайте их в разделе комментариев ниже.
| Ссылка: | Прогнозирование рака молочной железы с помощью Apache Spark Машинное обучение Логистическая регрессия от нашего партнера JCG Кэрол Макдональд в блоге Mapr . |