Во вторник, в конце моего 5-часового ускоренного курса по машинному обучению для актуариев, Пьер задал мне интересный вопрос о времени вычислений различных методов. Я представлял философию различных алгоритмов, но я забыл упомянуть время вычислений. Я хотел попробовать несколько алгоритмов классификации на наборе данных, используемых для иллюстрации методов
> rm(list=ls())
> myocarde=read.table(
"http://freakonometrics.free.fr/myocarde.csv",
head=TRUE,sep=";")
> levels(myocarde$PRONO)=c("Death","Survival")Но набор данных довольно мал, с 71 наблюдением и 7 объяснительными переменными. Поэтому я решил повторить наблюдения и добавить несколько ковариат,
> levels(myocarde$PRONO)=c("Death","Survival")
> idx=rep(1:nrow(myocarde),each=100)
> TPS=matrix(NA,30,10)
> myocarde_large=myocarde[idx,]
> k=23
> M=data.frame(matrix(rnorm(k*
+ nrow(myocarde_large)),nrow(myocarde_large),k))
> names(M)=paste("X",1:k,sep="")
> myocarde_large=cbind(myocarde_large,M)
> dim(myocarde_large)
[1] 7100 31
> object.size(myocarde_large)
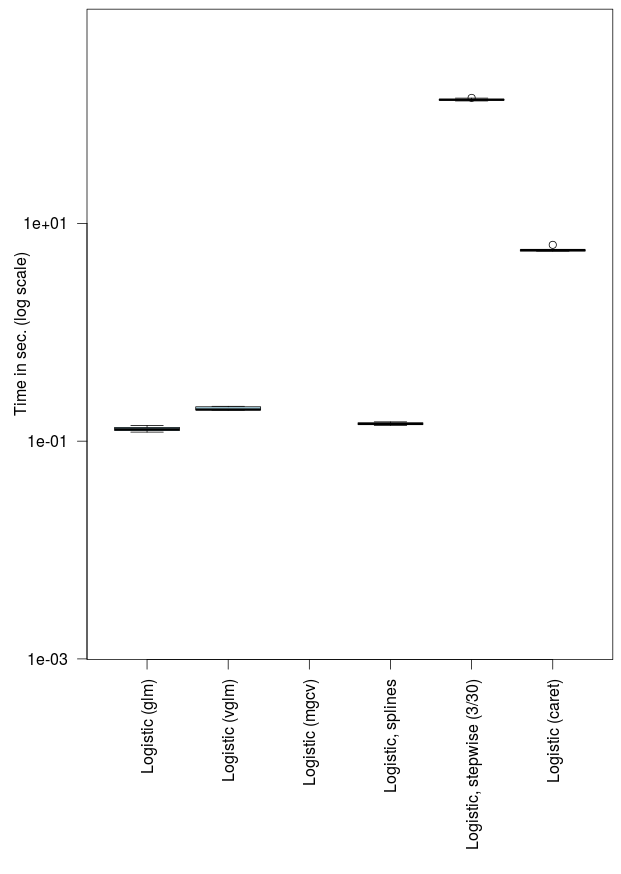
2049.064 kbytesНабор данных не большой … но, по крайней мере, он не занимает 0,0001 сек. запустить регрессию. На самом деле, чтобы запустить логистическую регрессию , требуется 0,1 секунды
> system.time(fit< glm(PRONO~.,
+ data=myocarde_large, family="binomial"))
user system elapsed
0.114 0.016 0.134
> object.size(fit)
9,313.600 kbytesИ я был удивлен, что объект регрессии был 9Mo, что более чем в четыре раза превышает размер набора данных. С большим набором данных, в 100 раз больше,
> dim(myocarde_large_2)
[1] 710000 31это занимает 20 сек.
> system.time(fit<-glm(PRONO~.,
+ data=myocarde_large_2, family="binomial"))
utilisateur système écoulé
16.394 2.576 19.819
> object.size(fit)
90,9025.600 kbytesи объект «только» в десять раз больше.
Обратите внимание, что со сплайном время вычислений довольно схоже
> library(splines)
> system.time(fit<-glm(PRONO~bs(INSYS)+.,
+ data=myocarde_large, family="binomial"))
user system elapsed
0.142 0.000 0.143
> object.size(fit)
9663.856 kbytesЕсли мы используем другую функцию, точнее ту, которую я использую для полиномиальных регрессий, она будет в два раза длиннее
> library(VGAM)
> system.time(fit1<-vglm(PRONO~.,
+ data=myocarde_large, family="multinomial"))
user system elapsed
0.200 0.020 0.226
> object.size(fit1)
6569.464 kbytesв то время как объект меньше. Теперь, если мы используем пошаговую процедуру в обратном направлении, она будет немного длиннее: почти на одну минуту, в 500 раз дольше, чем одна логистическая регрессия.
> system.time(fit<-step(glm(PRONO~.,data=myocarde_large,
family="binomial")))
...
Step: AIC=4118.15
PRONO ~ FRCAR + INCAR + INSYS + PRDIA + PVENT + REPUL + X16
Df Deviance AIC
<none> 4102.2 4118.2
- X16 1 4104.6 4118.6
- PRDIA 1 4113.4 4127.4
- INCAR 1 4188.4 4202.4
- REPUL 1 4203.9 4217.9
- PVENT 1 4215.5 4229.5
- FRCAR 1 4254.1 4268.1
- INSYS 1 4286.8 4300.8
user system elapsed
50.327 0.050 50.368
> object.size(fit)
6,652.160 kbytesЯ тоже хотел попробовать карет. В этом пакете приятно сравнивать модели. В обзоре книги « Вычислительная актуарная наука с R» в JRSS-A Андрей Костеко заметил, что этот пакет даже не упоминался и его не было. Поэтому я попробовал логистическую регрессию
> library(caret)
> system.time(fit<-train(PRONO~.,
+ data=myocarde_large,method="glm"))
user system elapsed
5.908 0.032 5.954
> object.size(fit)
12,676.944 kbytesЭто заняло 6 секунд (в 50 раз больше, чем стандартный вызов функции glm), и объект довольно большой. Это даже хуже, если мы попытаемся выполнить пошаговую процедуру
> system.time(fit<-train(PRONO~.,
+ data=myocarde_large,method="glmStepAIC"))
...
Step: AIC=4118.15
.outcome ~ FRCAR + INCAR + INSYS + PRDIA + PVENT + REPUL + X16
Df Deviance AIC
<none> 4102.2 4118.2
- X16 1 4104.6 4118.6
- PRDIA 1 4113.4 4127.4
- INCAR 1 4188.4 4202.4
- REPUL 1 4203.9 4217.9
- PVENT 1 4215.5 4229.5
- FRCAR 1 4254.1 4268.1
- INSYS 1 4286.8 4300.8
user system elapsed
1063.399 2.926 1068.060
> object.size(fit)
9,978.808 kbytesчто заняло 15 минут, всего 30 ковариат … Вот график (я использовал микробенчмарк для его построения)
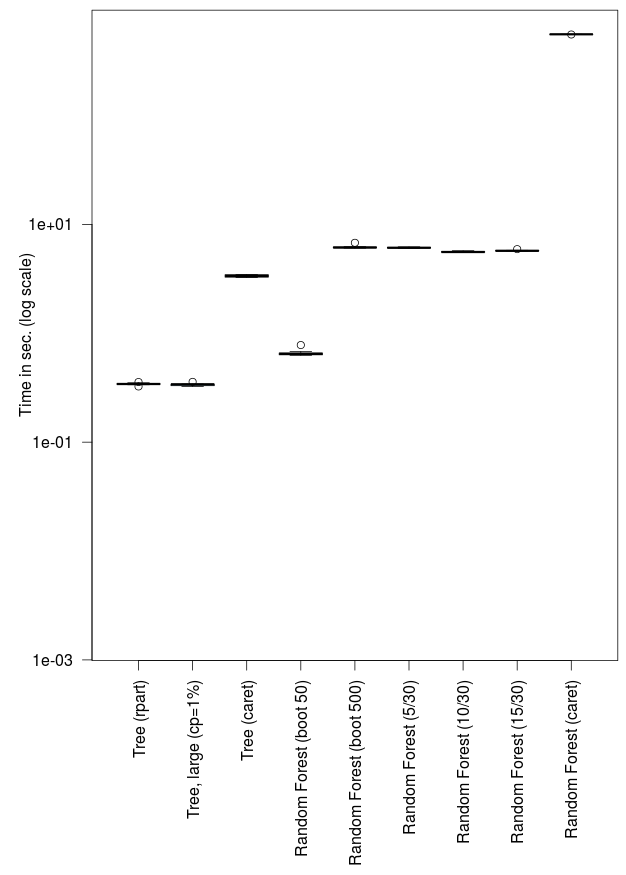
> library(rpart)
> system.time(fit<-rpart(PRONO~.,
+ data=myocarde_large))
user system elapsed
0.341 0.000 0.345
> object.size(fit4)
544.664 kbytesЗдесь это быстро, и объект довольно маленький. И если мы изменим параметр сложности, чтобы получить более глубокое дерево, это почти то же самое
> system.time(fit<-rpart(PRONO~.,
+ data=myocarde_large,cp=.001))
user system elapsed
0.346 0.000 0.346
> object.size(fit)
544.824 kbytesНо опять же, если мы запустим ту же функцию через каретку, она будет более чем в десять раз медленнее,
> system.time(fit<-train(PRONO~.,
+ data=myocarde_large,method="rpart"))
user system elapsed
4.076 0.005 4.077
> object.size(fit)
5,587.288 kbytesи объект в десять раз больше. Теперь рассмотрим случайный лес .
> library(randomForest)
> system.time(fit<-randomForest(PRONO~.,
+ data=myocarde_large,ntree=50))
user system elapsed
0.672 0.000 0.671
> object.size(fit)
1,751.528 kbytesПри «только» 50 деревьях получить результат можно только в два раза дольше. Но с 500 деревьями (значение по умолчанию) требуется в двадцать раз больше (при разумном пропорциональном времени выращивание 500 деревьев вместо 50)
> system.time(fit<-randomForest(PRONO~.,
+ data=myocarde_large,ntree=500))
user system elapsed
6.644 0.180 6.821
> object.size(fit)
5,133.928 kbytesЕсли мы изменим число используемых ковариат, на каждом узле мы увидим, что это почти не влияет. С 5 ковариатами (что является квадратным корнем от общего числа ковариат, т.е. это значение по умолчанию), это занимает 6 секунд,
> system.time(fit<-randomForest(PRONO~.,
+ data=myocarde_large,mtry=5))
user system elapsed
6.266 0.076 6.338
> object.size(fit)
5,161.928 kbytesно если мы используем 10, это почти то же самое (даже меньше)
> system.time(fit<-randomForest(PRONO~.,
+ data=myocarde_large,mtry=10))
user system elapsed
5.666 0.076 5.737
> object.size(fit)
2,501.928 bytesЕсли мы используем алгоритм случайного леса внутри каретки, это займет 10 минут,
> system.time(fit<-train(PRONO~.,
+ data=myocarde_large,method="rf"))
user system elapsed
609.790 2.111 613.515и визуализация
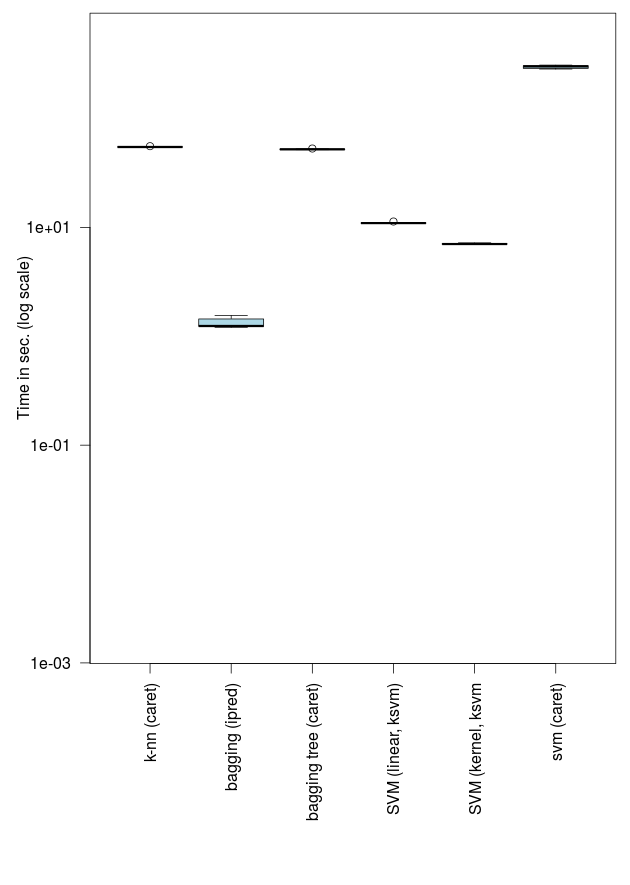
Если мы рассмотрим метод k- ближайшего соседа, с осторожностью снова, это займет некоторое время, снова с 10 минутами
> system.time(fit<-train(PRONO~.,
+ data=myocarde_large,method="knn"))
user system elapsed
66.994 0.088 67.327
> object.size(fit)
5,660.696 kbytesэто почти то же самое время, что и алгоритм упаковки, на деревьях
> system.time(fit<-train(PRONO~.,
+ data=myocarde_large,method="treebag"))
Le chargement a nécessité le package : plyr
user system elapsed
60.526 0.567 61.641
> object.size(fit)
72,048.480 kbytesно на этот раз объект довольно большой!
Мы также можем рассмотреть методы SVM со стандартным евклидовым расстоянием
> library(kernlab)
> system.time(fit<-ksvm(PRONO~.,
+ data=myocarde_large,
+ prob.model=TRUE, kernel="vanilladot"))
Setting default kernel parameters
user system elapsed
14.471 0.076 14.698
> object.size(fit)
801.120 kbytesили используя какое-то ядро
> system.time(fit<-ksvm(PRONO~.,
+ data=myocarde_large,
+ prob.model=TRUE, kernel="rbfdot"))
user system elapsed
9.469 0.052 9.701
> object.size(fit)
846.824 kbytesОба метода занимают около 10 секунд, намного больше, чем наша основная логистическая регрессия (в сто раз больше). И снова, если мы попытаемся использовать каретку, чтобы сделать то же самое, это займет некоторое время …
> system.time(fit<-train(PRONO~.,
+ data=myocarde_large, method="svmRadial"))
user system elapsed
360.421 2.007 364.669
> object.size(fit)
4,027.880 kbytesВывод следующий
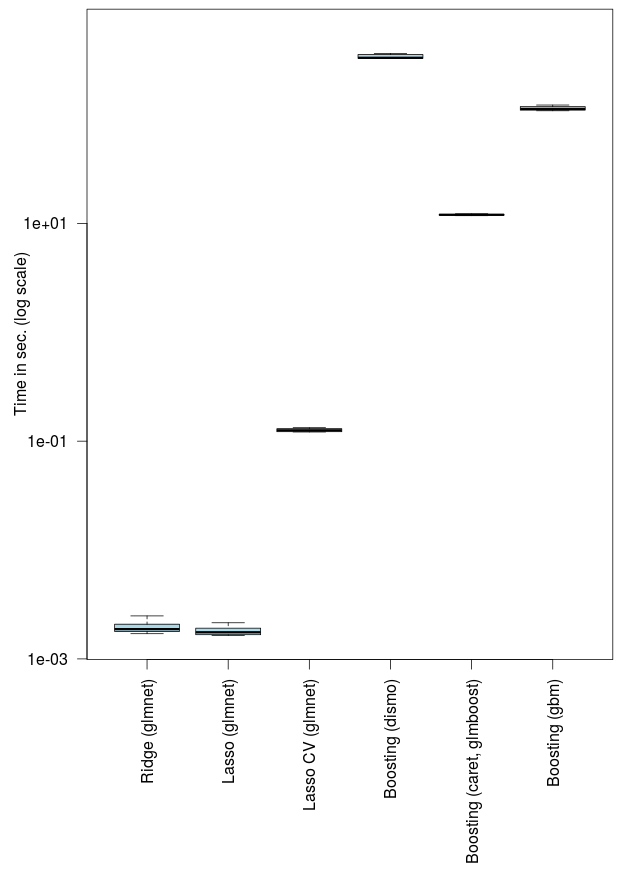
Я также хотел попробовать некоторые функции, такие как Ridge и LASSO.
> library(glmnet)
> idx=which(names(myocarde_large)=="PRONO")
> y=myocarde_large[,idx]
> x=as.matrix(myocarde_large[,-idx])
> system.time(fit<-glmnet(x,y,alpha=0,lambda=.05,
+ family="binomial"))
user system elapsed
0.013 0.000 0.052
> system.time(fit<-glmnet(x,y,alpha=1,lambda=.05,
+ family="binomial"))
user system elapsed
0.014 0.000 0.013Я был удивлен, увидев, как быстро это. И если мы используем перекрестную проверку для количественной оценки штрафа
> system.time(fit10<-cv.glmnet(x,y,alpha=1,
+ type="auc",nlambda=100,
+ family="binomial"))
user system elapsed
11.831 0.000 11.831Это занимает некоторое время … но это разумно, по сравнению с другими методами. И, наконец, рассмотрим несколько улучшающих пакетов.
> system.time(fit<-gbm.step(data=myocarde_large,
+ gbm.x = (1:(ncol(myocarde_large)-1))[-idx],
+ gbm.y = ncol(myocarde_large),
+ family = "bernoulli", tree.complexity = 5,
+ learning.rate = 0.01, bag.fraction = 0.5))
user system elapsed
364.784 0.428 365.755
> object.size(fit)
8,607.048 kbytesЭто было долго. Более 6 минут. Использование пакета glmboost через каретку было намного быстрее, на этот раз
> system.time(fit<-train(PRONO~.,
+ data=myocarde_large,method="glmboost"))
user system elapsed
13.573 0.024 13.592
> object.size(fit)
6,717.400 bytesПри использовании GBM через каретку было в десять раз дольше,
> system.time(fit<-train(PRONO~.,
+ data=myocarde_large,method="gbm"))
user system elapsed
121.739 0.360 122.466
> object.size(fit)
7,115.512 kbytes
Все это было сделано одним ноутбуком. Теперь мне нужно запустить те же коды на более быстрой машине, чтобы попробовать гораздо большие наборы данных….